Lab Etiquette: Difference between revisions
No edit summary |
No edit summary |
||
| (9 intermediate revisions by the same user not shown) | |||
| Line 1: | Line 1: | ||
'''Diese Seite ist auch auf [[Lab Etiquette (ENG)|Englisch]] verfügbar.''' | '''Diese Seite ist auch auf [[Lab Etiquette (ENG)|Englisch 🇬🇧]] verfügbar.''' | ||
== | == '''Laborzugang''' == | ||
Das Laborgebäude ML ist zwischen 6:30 und 20:30 Uhr an Werktagen geöffnet. Zugang zu den Laborräumen erhält man mittels RFID-Transponder, der es auch erlaubt, das Laborgebäude außerhalb der oben stehenden Öffnungszeiten zu betreten. Masterstudenten und Doktoranden erhalten einen persönlichen Transponder über die Schlüsselausgabe (Formular bei Petra Schnurr) und müssen den Transponder nach Beendigung ihrer Zeit als Mitarbeiter wieder abgeben. Für Bachlorstudenten und Hiwis gibt es nach Einarbeitung in Ausnahmefällen einen Leihtransponder für notwendige Arbeiten außerhalb der Öffnungszeiten. Der Leihtransponder befindet sich im Schlüsselkasten am allg. Computer-Arbeitsplatz. Die Entnahme des Leihtransponders ist in der Liste mit Datum und Kürzel zu vermerken. | |||
== | == '''Arbeitszeiten''' == | ||
Nicht-wissenschaftliche Mitarbeiter und Hiwis sind verpflichtet, die Arbeitszeit zu erfassen. Hierzu steht den Mitarbeitern die Online-Webseite der Personalverwaltung zur Verfügung: | |||
''<nowiki>https://personal.uni-konstanz.de/qisserver/rds?state=user&type=0&topitem=</nowiki>'' | |||
Hiwis nutzen zur Zeiterfassung den Stundenzettel (erhältlich bei Petra Schnurr) | |||
Wissenschaftliche Mitarbeiter sollen zu den Kernarbeitszeiten 9.00 – 12.00 und 14.00 – 16.00 anwesend sein. Eine Abwesenheit ist mit Christof abzusprechen und im Wandkalender im Eingangsbereich der Bürozeile zu vermerken. | |||
== '''Krankheit''' == | |||
Im Krankheitsfall soll so bald wie möglich Christof und das Sekretariat per Telefon und/oder E-Mail benachrichtigt werden, damit Vertretung, etc. organisiert werden kann. Bei längerer Krankheit muss am dritten Tag der Abwesenheit eine ärztliche Krankmeldung/ein ärztliches Attest an petra.schnurr@uni-konstanz.de geschickt werden. | |||
== '''Urlaub''' == | |||
Mitarbeiter beantragen möglichst frühzeitig (mindestens 1 Woche vorher) ihren Urlaub über das online Zeiterfassungssystem: | |||
''<nowiki>https://personal.uni-konstanz.de/qisserver/rds?state=user&type=0&topitem=</nowiki>'' | |||
Die Möglichkeit für längerfristigen Urlaub (> 2 Wochen) ist projektabhängig und soll mindestens 2 Monate vorher mit Christof abgesprochen werden. In der 2.Hälfte des Wintersemesters (Anfang Januar – Ende Februar) ist während des VTK Urlaub nur in Ausnahmefällen möglich. | |||
Vor Urlaubsantritt muss immer die Kultur/Lagerung von wichtigen Klonen/Zellinien/Proben sichergestellt und eine Vertretung für den Labjob organisiert sein. Bachelor-/Masterstudenten sprechen eine mehrtägige Abwesenheit mit Ihrem/Ihrer BetreuerIn und Christof ab. | |||
== '''Seminare/Projektbesprechungen''' == | |||
Montags vormittags (8.45 Uhr) ist eine allg. Laborbesprechung. Im Anschluß daran eine Porjektvorstellung einzelner Mitarbeiter, die am Notice-Board im Eingangsbereich angekündigt wird. | |||
Mittwochs am Nachmittag von 15.15 Uhr – ca. 17.00 Uhr findet unser Journal-Club statt. Die jeweils behandelte Publikation wird in der Woche zuvor auf den Fileserver geladen bzw. als pdf-file an alle verschickt. Im 14-tägigen Turnus besprechen wir in diesem Seminar aktuelle neue Daten aller Mitarbeiter. | |||
Die Teilnahme an diesen beiden Seminaren ist obligatorisch, der Seminarplan wird in der Regel auf ein viertel- bis ein halbes Jahr im voraus festgelegt. | |||
Dienstags nachmittags und donnerstags zur Mittagszeit finden während der Vorlesungszeit die Seminare der Graduiertenschule KoRS-CB sowie das Fachbereichsseminar des FB Biologie statt. Die Vortragenden/Themen werden durch Aushang bekannt gegeben. Die Teilnahme ist fakultativ, aber wenn möglich sollte man sich die interessanten Vorträge nicht entgehen lassen. | |||
Spezifische Projektbesprechungen finden nach Absprache mit Christof statt. Zu den Projektbesprechungen ist das Laborbuch als Gesprächsgrundlage mitzubringen. | |||
== '''Namenskürzel/Beschriftungen''' == | |||
Jeder Mitarbeiter benutzt ein unverwechselbares Namenskürzel, mit dem alle Proben, Lösungen, etc. beschriftet werden müssen. Die Namenskürzel werden im Sekretariat vergeben und sind in einer Liste auf dem Fileserver abgelegt: | |||
Fileserver_LSH/Vorlagen/Labor-Verzeichnisse/Namenskuerzel_AG Hauck_2025.doc | |||
== '''Laborbuch''' == | |||
Jeder Mitarbeiter führt ein Laborbuch, das im Sekretariat ausgegeben wird und in dem alle Arbeiten in chronologischer Reihenfolge dokumentiert werden. Muster für die Dokumentation von verschiedenen Experimenten sind als pdf-file auf dem Fileserver abgelegt: | |||
/ | Fileserver_LSH/Vorlagen/Labor-Protokollierung/ | ||
* Gentamicin-Assay: „Steffi“ | |||
* PCR: „''PCR-based-LIC-cloning-protocol-CRH.pdf''“ | |||
* Zelltransfektion: „''HEKcell-transfection-Protocol-JWK.pdf''“ | |||
* In vitro Kinase Assay: “Erik” | |||
* [[Immunoprecipitation|Immunopräzipitation]] (MHE) | |||
* [[Protein expression and purification]]: “Sarah” | |||
* Lentivirus production: “Marlene” | |||
* Bacterial growth curve: “Ann-Kathrin” | |||
Die Seiten des Laborbuchs werden durchnumeriert und auf den ersten beiden Seiten wird ein Inhaltsverzeichnis angelegt. | |||
Das Laborbuch gehört dem Lehrstuhl Zellbiologie und verbleibt nach dem Ausscheiden im Besitz des Lehrstuhls. Aus dem Laborbuch dürfen keine Seiten entfernt werden. Originalausdrucke, Kopien von Gelen, Röntgenfilme, etc. werden beschriftet und dem Laborbuch beigelegt, indem sie zu den entsprechenden Experimenten geheftet oder geklebt werden. Die elektronischen Originaldaten werden auf dem Fileserver in einem eigenen User-Verzeichnis gespeichert (s. Kapitel Daten Archivierung). | |||
Für die meisten Experimente gibt es bereits Labor-Protokolle, die in unserem VTK-Skript ausführlich beschrieben sind und auf die im Laborbuch verwiesen werden kann. Das VTK-Skript findet sich auf dem Fileserver: | |||
''Fileserver_LSH/Vorlagen/Labor-Protokollierung/Skript-Vertiefungskurs-2025.doc'' | |||
Die verwendeten Reagenzien und Lösungen sind ebenfalls entweder in der Rezepte-Datei beschrieben: ''Fileserver_LSH/Rezepte/Rezepte-2025.doc'' oder sind in der Chemikalienliste zu finden: | |||
''Fileserver_LSH/Chemikalienliste/Chemikalienliste 2025 Gruppe Hauck.xls'' | |||
<u>Nur bei Abweichen vom existierenden Protokoll sind besondere Vermerke im Laborbuch notwendig.</u> | |||
Bei mikroskopischen Arbeiten, durchflußzytometrischen Analysen oder ähnlichen Arbeiten mit Ergebnissen in elektronischer Form wird ein Probenprotokoll angefertigt (s. Fileserver_LSH/Vorlagen/Labor-Protokollierung/Protokoll-Mikroskop-FACS.doc“) und dem Experiment beigeheftet. | |||
Bei Projektbesprechungen dient das Laborbuch als Grundlage für die Diskussion der Ergebnisse und ist deshalb unbedingt mitzubringen. | |||
== '''Daten-Archivierung''' == | |||
Standardexperimente sollen anhand der oben angeführten Laborprotokolle durchgeführt werden, Abweichungen sind im Laborbuch festzuhalten. Die im Rahmen der Experimente erhaltenen Rohdaten (z.B. Mikroskopiebilder, Gelbilder, FACS-Daten, VarioScan-Daten) sowie die daraus hergestellten abgeleiteten Abbildungen (Graphiken, berbeiteten Mikroskopiebilder, etc.) sind auf dem Daten-Fileserver „Fileserver_LSH_Data/00_User/„ in einem separaten Nutzerfolder zu speichern. Nur dort erfolgt eine automatische (tägliche) Sicherung, so dass keine Daten verloren gehen können. | |||
Jede elektronische Datendatei trägt im Namen das Datum, das Namenskürzel und das Experiment (The raw data files are named according to the following scheme): | |||
'''Jahr-Monat-Tag_Namenskürzel_Experiment / year-month-day_initials_experiment''' | |||
''' | |||
z.B. /e.g.: 2025-01-01_CRH_Adhesion assay | |||
- the digital version/raw files should be on the file server with the date that can also be found in the lab book | |||
- a printed version of the evaluated data should be attached in the lab book | |||
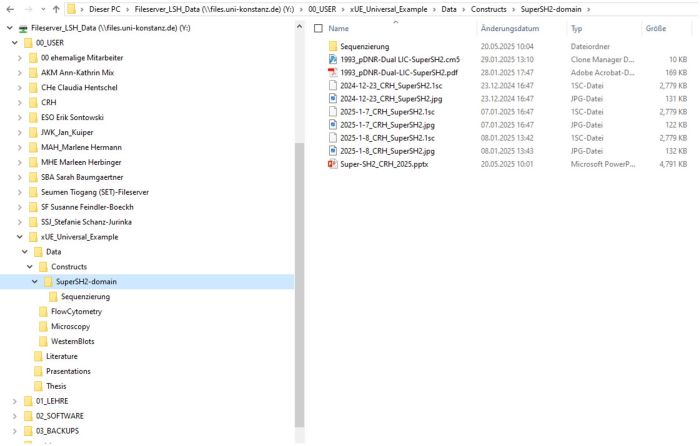
For later use in publications it is important that we all stick to the same folder structure and naming of files. An example of such a user folder is given on the Fileserver and depicted on the next page: | |||
Fileserver_LSH_Data/00_User/xUE_Universal_Example/ | |||
''' | '''Data''' | ||
'' Constructs'' | |||
<small>SuperSH2 agarose gels, sequencing results, plasmid map, etc.</small> | |||
'' Western Blots'' | |||
'' Immunofluorescence staining'' | |||
'' Flow Cytometry'' | |||
'' ELISA/SEAP-Assays'' | |||
''' .''' | |||
''' .''' | |||
'''Literature''' | |||
'''Presentations''' | |||
'''Thesis:''' for your thesis: for '''every''' figure in your thesis create a subfolder named with the Figure-Number and include in each subfolder both: the final figure as pdf- and illustrator-file and the '''used raw data''' (subdivided in panels A, B, etc.). The name of the raw data file should remain the same as it is found in your DATA-folder and lab book) | |||
[[File:Labetiquette fileserver.jpg|center|thumb|700x700px|'''Fig. 1:''' Example of the user folder structure on the Fileserver_LSH_Data of user UE_Universal_Example.]] | |||
== | == '''Laborarbeitsplatz''' == | ||
In den Laboren gibt es allgemeine Laborarbeitsplätze (z.B. in den beiden Zellkulturlaboren, im Infektionsraum oder der Bereich mit Proteingel-Elektrophorese) sowie private Arbeitsplätze, die zugewiesen sind. Arbeiten auf den privaten Arbeitsplätzen anderer soll nur nach Rücksprache erfolgen. Jeder ist für die Sauberhaltung seines Arbeitsplatzes, der dazugehörigen Regale und Schubladen selbst verantwortlich. Die Arbeitsfläche wird mindestens einmal pro Woche abgewischt und desinfiziert, die Schubladen/Regale sind regelmäßig abzustauben und ordentlich zu halten. | |||
Zusätzlich hat jeder einen Lagerplatz in einem Kühlschrank bei 4°C, in einem Gefrierschrank/-fach bei -20°C und kann Proben in beschrifteten Boxen im -80°C-Schrank in Raum ML lagern. Für die Lagerung stehen Kunststoffboxen mit Deckel zur Verfügung, offenen Ständer (speziell für Eppendorfgefäße) sollen nur für Kurzzeit-Aufbewahrung (1 Tag) verwendet werden. Alle Proben (Eppis, Agarplatten, etc.) müssen mit Kürzel, Inhaltsbezeichnung und Datum beschriftet sein. Alle Lagerplätze sollen regelmäßig (mindestens 1x je Monat) aufgeräumt/aktualisiert werden. Bei Lagerung im -80°C Schrank ist für jede Box ein Datenblatt in den -80°C-Ordner abzulegen. Eine Vorlage für das Datenblatt findet sich auf dem Fileserver: | |||
''Fileserver_LSH/Vorlagen/Labor-Protokollierung/'' ''-80C Box-Vorlage.doc'' | |||
Das Öffnen von Tiefgefrierschränken bitte auf ein Minimum reduzieren (da sonst Vereisung und Fehlfunktion). Deshalb bereits VOR dem Öffnen überlegen, was entnommen werden soll und wo es zu finden ist (s. Datenblatt!!!). Allgemeine Laborarbeitsplätze sind nach jeder Benutzung zu säubern (z.B. sterile Werkbank, Proteingel-Elektrophorese, Abzug, Waagen, Geldoku) und alle verwendeten Lösungen, Apparaturen, etc. müssen wieder aufgeräumt werden. | |||
== '''Lab-Jobs''' == | |||
Jeder Mitarbeiter erhält einen Labjob, den er für die Allgemeinheit wahrnimmt. Der Lab-Job kann die Pflege einer allgemeinen Sammlung, die Verantwortung für ein Gerät oder die Aufsicht und Organisation eines Labors beinhalten. Darüber hinaus wird die Herstellung von Lösungen/Puffern etc. für die Allgemeinheit über Lab-Jobs. Die jeweils Verantwortlichen sind auf einer Liste am am Noticeboard am Eingang aufgeführt. Die Raum-Verantwortlichen organisieren auch die regelmäßige Pflege/Putzpläne für allgemeine Geräte wie sterile Werkbänke, Inkubatoren, etc. | |||
=== | == '''Schließdienst''' == | ||
Der abendliche Schließdienst, der dafür sorgt, dass Geräte wie sterile Werkbänke, Vakkumpumpen, Geldoku oder die Propangasleitung ausgeschaltet sind, wechselt im wöchentlichen Turnus unter den wissenschaftlichen Mitarbeitern und Masterstudenten. Eine Liste für den abendlichen Kontrollgang des Schließdienstes hängt am Noticeboard am Eingang. | |||
== '''Computernutzung''' == | |||
Im Seminarraum befindet sich ein Computer zur allgemeinen Nutzung. Bitte jederzeit die Nutzung für dienstliche Zwecke ermöglichen. Private PCs/Notebooks können an das Netzwerk angeschlossen werden. Voraussetzung ist ein aktueller Virenscanner und die Aktivierung einer Firewall. Den Zugang zu den Netzwerk-Speichern des Lehrstuhls im Rechenzentrum (Fileserver_LSH und Fileserver_LSH_Data) richtet Christof ein. Auf dem Fileserver_LSH_Data/02_SOFTWARE stehen verschiedene Software-Programme zur Verfügung, darunter Adobe Photoshop CS4, Adobe Acrobat Pro9, Adobe Illustrator CS4, MS Office 2016, Clone Manager 9, Endnote X9, SigmaStat 4.0, Leica LAS, Biorad QuantityOne, VarioScan SkanIt. Um den problemlosen Austausch der Daten innerhalb des Labors zu gewährleisten, müssen die oben angegebenen Versionen dieser Programme für alle dienstlichen Zwecke verwendet werden bzw. die Daten in einer mit diesen Programmversionen kompatiblen Form gespeichert werden. | |||
== '''Die Mitarbeit jedes Einzelnen ist gefragt''' == | |||
Jeder Mitarbeiter kann durch wenig Aufwand dazu beitragen, dass sich die Zusatzarbeiten im Labor für Alle minimieren. So soll jeder durch vorausschauende Planung den anfallenden Verbrauch/Abfall/Aufräumaufwand möglichst gering halten und auch bei der Entsorgung von Abfällen mithelfen. Bitte Autoklaviersäcke nicht bis zum „Überquellen“ füllen, sondern rechtzeitig verschließen, in der Wanne deponieren und neuen Autoklaviersack in den entsprechenden Müllbehälter geben. | |||
Jeder ist auch selbst für das Befüllen der Boxen mit Pipettenspitzen und das Auffüllen von Eppis verantwortlich. Autoklaviergut wird in der Plastikkiste gesammelt und von Claudia/Susanne für alle autoklaviert. | |||
Zum verträglichen Umgang gehört auch das Verhalten und die sauberkeit in der Teeküche und im Sozialraum. Wenn ich im Verlaufe von 1 Monat nie die Spülmaschine ein- oder ausgeräumt oder die Kaffeemaschine aufgefüllt habe, dann habe ich mich sehr wahrscheinlich auf Kosten von Anderen am sauberen Geschirr bedient. | |||
== '''Chemikalien/Lösungen''' == | |||
Viele Lösungen werden als Lab-Job angesetzt und entweder als Stocklösung an festgelegten Plätzen für Alle zugänglich aufbewahrt oder als aliquotierte Lösung bereitgestellt, wobei man sich bei Bedarf ein Aliquot an den eigenen Arbeitsplatz mitnimmt und dort für die weitere Verwendung aufbewahrt. Weitere Lösungen für den eigenen Bedarf sind selbst anzusetzen (bitte die benötigten Mengen im Voraus abschätzen und nicht Liter-weise Lösungen ansetzen, wenn man für zwei Experimente insgesamt nur 10 ml braucht) und korrekt beschriftet (Kürzel, Datum, Inhalt: Bezeichnung und Konzentration; Gefahrensymbol) am eigenen Arbeitsplatz aufzubewahren. Lösungen und Aliquots am Arbeitsplatz der Kollegen NUR nach Rücksprache benutzen. Stocklösungen, aliquotierte Lösungen und die jeweiligen Verantwortlichen sind auf der Liste am Schwarzen Brett „Lab-Jobs“ aufgeführt. Für das Ansetzen von Lösungen gibt es ein Rezeptbuch, das als Datei auf dem Fileserver bereit liegt: | |||
Fileserver_LSH//Rezepte/ Rezepte-2025.doc | |||
== '''Allgemeine Sammlungen''' == | |||
Der Lehrstuhl unterhält verschiedene Sammlungen, die eine wichtige Ressource für Alle darstellen und mit denen deshalb besonders umsichtig und verantwortlich umgegangen werden muss. | |||
<u>Stammsammlung:</u> umfasst alle Bakterienstämme, die nach Nummern abgelegt sind. Lagerung bei -70°C in Truhe auf Ebene 5 sowie in Raum ML6 : | |||
– nichtpathogene Bakterien (=gleichzeitig Repositorium aller hergestellten Plasmide) | |||
– pathogene Bakterien und Mutanten (Schlüssel dafür bei Christof) | |||
Die Stammsammlung ist auch als MS Access-Datei vorhanden. Jeder neu hergestellte oder von extern erhaltene Bakterienstamm erhält eine neue Stammnummer und wird in der Stammsammlung eingefroren sowie elektronisch erfasst. Die Verteilung der Nummern erfolgt über Christof. Zugang zur Stammsammlung haben nur Susanne und Christof. | |||
<u>Plasmidsammlung:</u> umfasst alle isolierten Plasmide aus den nichtpathogenen Bakterienstämmen der Stammsammlung. Die Numerierung ist identisch mit der Stammsammlung (Lagerung im allg. Kühlschrank bei 4°C) | |||
<u>Zellsammlung:</u> umfasst Stocks aller Zellinien und primärer Zellen. Werden im flüssigen Stickstoff in der Medienküche aufbewahrt. Die Zellsammlung wird von Susanne verwaltet. | |||
<u>Virensammlung:</u> umfasst alle lentiviralen, adenoviralen, AAV-Partikel und Bakteriophagen. Werden bei -70°C aufbewahrt. Access-Datei! | |||
<u>Oligosammlung:</u> umfasst alle Oligos, die für Klonierungen, PCRs, etc. bestellt wurden. Die Oligos werden als 10 mM Stammlösung in TE-Puffer bei -20°C in der allgemeinen Sammlung, nicht in privaten Gefrierschränken, aufbewahrt. Bestellung neuer Oligos und Ablage der Informationen erfolgt über Jan Kuiper. | |||
<u>Antikörper-Sammlung:</u> Sammlung aller Primärantikörper (kommerziell oder selbst hergestellt) und Sekundärantikörper (Enzym- oder Fluoreszenz-konjugiert). Datenblätter werden im Ordner „Antikörper“ gesammelt. Claudia Hentschel organisiert die Antikörper-Sammlung. | |||
<u>Protein-Sammlung:</u> Hier werden alle rekombinanten, gereinigten Proteine erfasst. | |||
<u>Inhibitor-Sammlung:</u> Hier werden Informationen zu allen pharmakologischen Inhibitoren und die Konzentration, das Lösungsmittel und der Aufbewahrungsort von Stammlösungen abgelegt. _______________ organisiert die Inhibitor-Sammlung. | |||
<u>Enzym-Sammlung:</u> umfasst alle kommerziellen und selbst hergestellten (DpnI) Restriktionsenzyme, die bei -20°C gelagert werden. Entnahme von Enzymen aus den Vorratsgefäßen nur mit FILTERSPITZEN! | |||
Die Enzyme dürfen nur für eine möglichst kurze Zeitspanne (1-2 Minuten) aus dem Gefrierschrank entnommen werden (d.h. direkt vor Gebrauch) und werden während dieser kurzen Zeit in einem auf -20°C-gekühlten Paraffinblock gelagert. | |||
''Ordner mit Dokumentation zu allen Sammlungen finden sich in den Regalen am allgemeinen Arbeitsplatz im Bürobereich.'' | |||
== '''Kompetente Zellen''' == | |||
Aliquots kompetenter Bakterien werden bei -80°C gelagert. Je Aliquot sind 200 µl weggefroren, ausreichend für 2 Transformationen. Verschiedene Stämme stehen für unterschiedliche Zwecke zur Verfügung. Der Standardklonierungsstamm ist NovaBlue. Bei der Entnahme möglichst zügig vorgehen, Erwärmung der Aliquots führt zu verringerter Kompetenz, nicht mehr Aliquots entnehmen als tatsächlich gebraucht werden. | |||
== '''Enzyme''' == | |||
Polymerasen, Restriktionsenzyme, etc. werden bei -20°C in Boxen bzw. auf Paraffinblöcken gemeinsam mit den entsprechenden Puffern aufbewahrt. Diese Enzyme werden von Allen gemeinsam benutzt. Deshalb besonders reinlich mit den Enyzm-Stammlösungen umgehen und Verunreinigungen (Filterspitzen !!!) sowie Erwärmung vermeiden. | |||
Bei der Entnahme von Enzymen, die Eppis mit der Stammlösung ausschließlich in einem auf -20°C-gekühlten Paraffinblöckchen zum Arbeitsplatz transportieren und darin zum Pipettieren belassen. Auch mit Paraffinblock möglichst das Enzym nur kurzzeitig aus dem -20°C Gefrierschrank entnehmen (< 2 Minuten) und sofort nach dem Pipettieren wieder zurückbringen. Beim Öffnen der Vorratsgefäße auf jeden Fall Kontakt der Deckelinnenseite mit der Haut vermeiden!! | |||
== '''Bestellungen''' == | |||
Wenn allgemeine Chemikalien oder Einwegartikel zur Neige gehen, RECHTZEITIG (d.h. vor dem Aufbrauchen der letzten Packung oder dem Abpipettieren des letzten Mikroliters) Bescheid geben: entweder Anschreiben an die Tafel im Labor oder den Bestellern sagen. Projektspezifische Chemikalien/Antikörper etc. oder Laborutensilien/-geräte vor Bestellung mit Christof absprechen. | |||
Für Oligobestellungen über Jan die Oligo-Informationen in die Bestell-Liste auf dem Fileserver eintragen: Fileserver_LSH:\Stammsammlung\Primer\01_PrimerOrdering_Primerbestellung | |||
Andere on-line Bestellungen mit Christof absprechen. | |||
Bestellung von Sequenzierungen über pre-paid labels von LGC oder GeneWiz. Bitte nicht mehr als 2 Sequenzierung pro neuem Konstrukt. Größeren Sequenzierbedarf mit Christof absprechen. Lieferungen von Reagenzien sollen dem Besteller übergeben werden. Besteller prüft auf Vollständigkeit (unter Nutzen des Lieferscheins) und bestätigt mit Datum und Unterschrift. Der Lieferschein und eventuell beiliegende Rechnungen werden im Sekretariat an Petra Schnurr weitergegeben. Bei Lieferung von Chemikalien, Antikörpern, Enzymen und anderen Reagenzien werden die Datenblätter mit einem Datum versehen und in den entsprechenden Ordnern abgelegt (s. „Allgemeine Sammlungen“). Auf den Datenblättern wird eventuell das Lösungsmittel, die Konzentration und eventuell abgefüllte Aliquots sowie der Lagerort vermerkt. | |||
ALLE Chemikalien, Reagenzien, Enzyme, etc. werden an zugänglichen und bekannten Orten gelagert. | |||
== '''Laborsicherheit''' == | |||
Siehe dazu [[All About Safety|Safety Guidelines]] | |||
Latest revision as of 14:22, 5 June 2025
Diese Seite ist auch auf Englisch 🇬🇧 verfügbar.
Laborzugang
Das Laborgebäude ML ist zwischen 6:30 und 20:30 Uhr an Werktagen geöffnet. Zugang zu den Laborräumen erhält man mittels RFID-Transponder, der es auch erlaubt, das Laborgebäude außerhalb der oben stehenden Öffnungszeiten zu betreten. Masterstudenten und Doktoranden erhalten einen persönlichen Transponder über die Schlüsselausgabe (Formular bei Petra Schnurr) und müssen den Transponder nach Beendigung ihrer Zeit als Mitarbeiter wieder abgeben. Für Bachlorstudenten und Hiwis gibt es nach Einarbeitung in Ausnahmefällen einen Leihtransponder für notwendige Arbeiten außerhalb der Öffnungszeiten. Der Leihtransponder befindet sich im Schlüsselkasten am allg. Computer-Arbeitsplatz. Die Entnahme des Leihtransponders ist in der Liste mit Datum und Kürzel zu vermerken.
Arbeitszeiten
Nicht-wissenschaftliche Mitarbeiter und Hiwis sind verpflichtet, die Arbeitszeit zu erfassen. Hierzu steht den Mitarbeitern die Online-Webseite der Personalverwaltung zur Verfügung:
https://personal.uni-konstanz.de/qisserver/rds?state=user&type=0&topitem=
Hiwis nutzen zur Zeiterfassung den Stundenzettel (erhältlich bei Petra Schnurr)
Wissenschaftliche Mitarbeiter sollen zu den Kernarbeitszeiten 9.00 – 12.00 und 14.00 – 16.00 anwesend sein. Eine Abwesenheit ist mit Christof abzusprechen und im Wandkalender im Eingangsbereich der Bürozeile zu vermerken.
Krankheit
Im Krankheitsfall soll so bald wie möglich Christof und das Sekretariat per Telefon und/oder E-Mail benachrichtigt werden, damit Vertretung, etc. organisiert werden kann. Bei längerer Krankheit muss am dritten Tag der Abwesenheit eine ärztliche Krankmeldung/ein ärztliches Attest an petra.schnurr@uni-konstanz.de geschickt werden.
Urlaub
Mitarbeiter beantragen möglichst frühzeitig (mindestens 1 Woche vorher) ihren Urlaub über das online Zeiterfassungssystem:
https://personal.uni-konstanz.de/qisserver/rds?state=user&type=0&topitem=
Die Möglichkeit für längerfristigen Urlaub (> 2 Wochen) ist projektabhängig und soll mindestens 2 Monate vorher mit Christof abgesprochen werden. In der 2.Hälfte des Wintersemesters (Anfang Januar – Ende Februar) ist während des VTK Urlaub nur in Ausnahmefällen möglich.
Vor Urlaubsantritt muss immer die Kultur/Lagerung von wichtigen Klonen/Zellinien/Proben sichergestellt und eine Vertretung für den Labjob organisiert sein. Bachelor-/Masterstudenten sprechen eine mehrtägige Abwesenheit mit Ihrem/Ihrer BetreuerIn und Christof ab.
Seminare/Projektbesprechungen
Montags vormittags (8.45 Uhr) ist eine allg. Laborbesprechung. Im Anschluß daran eine Porjektvorstellung einzelner Mitarbeiter, die am Notice-Board im Eingangsbereich angekündigt wird.
Mittwochs am Nachmittag von 15.15 Uhr – ca. 17.00 Uhr findet unser Journal-Club statt. Die jeweils behandelte Publikation wird in der Woche zuvor auf den Fileserver geladen bzw. als pdf-file an alle verschickt. Im 14-tägigen Turnus besprechen wir in diesem Seminar aktuelle neue Daten aller Mitarbeiter.
Die Teilnahme an diesen beiden Seminaren ist obligatorisch, der Seminarplan wird in der Regel auf ein viertel- bis ein halbes Jahr im voraus festgelegt.
Dienstags nachmittags und donnerstags zur Mittagszeit finden während der Vorlesungszeit die Seminare der Graduiertenschule KoRS-CB sowie das Fachbereichsseminar des FB Biologie statt. Die Vortragenden/Themen werden durch Aushang bekannt gegeben. Die Teilnahme ist fakultativ, aber wenn möglich sollte man sich die interessanten Vorträge nicht entgehen lassen.
Spezifische Projektbesprechungen finden nach Absprache mit Christof statt. Zu den Projektbesprechungen ist das Laborbuch als Gesprächsgrundlage mitzubringen.
Namenskürzel/Beschriftungen
Jeder Mitarbeiter benutzt ein unverwechselbares Namenskürzel, mit dem alle Proben, Lösungen, etc. beschriftet werden müssen. Die Namenskürzel werden im Sekretariat vergeben und sind in einer Liste auf dem Fileserver abgelegt:
Fileserver_LSH/Vorlagen/Labor-Verzeichnisse/Namenskuerzel_AG Hauck_2025.doc
Laborbuch
Jeder Mitarbeiter führt ein Laborbuch, das im Sekretariat ausgegeben wird und in dem alle Arbeiten in chronologischer Reihenfolge dokumentiert werden. Muster für die Dokumentation von verschiedenen Experimenten sind als pdf-file auf dem Fileserver abgelegt:
Fileserver_LSH/Vorlagen/Labor-Protokollierung/
- Gentamicin-Assay: „Steffi“
- PCR: „PCR-based-LIC-cloning-protocol-CRH.pdf“
- Zelltransfektion: „HEKcell-transfection-Protocol-JWK.pdf“
- In vitro Kinase Assay: “Erik”
- Immunopräzipitation (MHE)
- Protein expression and purification: “Sarah”
- Lentivirus production: “Marlene”
- Bacterial growth curve: “Ann-Kathrin”
Die Seiten des Laborbuchs werden durchnumeriert und auf den ersten beiden Seiten wird ein Inhaltsverzeichnis angelegt.
Das Laborbuch gehört dem Lehrstuhl Zellbiologie und verbleibt nach dem Ausscheiden im Besitz des Lehrstuhls. Aus dem Laborbuch dürfen keine Seiten entfernt werden. Originalausdrucke, Kopien von Gelen, Röntgenfilme, etc. werden beschriftet und dem Laborbuch beigelegt, indem sie zu den entsprechenden Experimenten geheftet oder geklebt werden. Die elektronischen Originaldaten werden auf dem Fileserver in einem eigenen User-Verzeichnis gespeichert (s. Kapitel Daten Archivierung).
Für die meisten Experimente gibt es bereits Labor-Protokolle, die in unserem VTK-Skript ausführlich beschrieben sind und auf die im Laborbuch verwiesen werden kann. Das VTK-Skript findet sich auf dem Fileserver:
Fileserver_LSH/Vorlagen/Labor-Protokollierung/Skript-Vertiefungskurs-2025.doc
Die verwendeten Reagenzien und Lösungen sind ebenfalls entweder in der Rezepte-Datei beschrieben: Fileserver_LSH/Rezepte/Rezepte-2025.doc oder sind in der Chemikalienliste zu finden:
Fileserver_LSH/Chemikalienliste/Chemikalienliste 2025 Gruppe Hauck.xls
Nur bei Abweichen vom existierenden Protokoll sind besondere Vermerke im Laborbuch notwendig.
Bei mikroskopischen Arbeiten, durchflußzytometrischen Analysen oder ähnlichen Arbeiten mit Ergebnissen in elektronischer Form wird ein Probenprotokoll angefertigt (s. Fileserver_LSH/Vorlagen/Labor-Protokollierung/Protokoll-Mikroskop-FACS.doc“) und dem Experiment beigeheftet.
Bei Projektbesprechungen dient das Laborbuch als Grundlage für die Diskussion der Ergebnisse und ist deshalb unbedingt mitzubringen.
Daten-Archivierung
Standardexperimente sollen anhand der oben angeführten Laborprotokolle durchgeführt werden, Abweichungen sind im Laborbuch festzuhalten. Die im Rahmen der Experimente erhaltenen Rohdaten (z.B. Mikroskopiebilder, Gelbilder, FACS-Daten, VarioScan-Daten) sowie die daraus hergestellten abgeleiteten Abbildungen (Graphiken, berbeiteten Mikroskopiebilder, etc.) sind auf dem Daten-Fileserver „Fileserver_LSH_Data/00_User/„ in einem separaten Nutzerfolder zu speichern. Nur dort erfolgt eine automatische (tägliche) Sicherung, so dass keine Daten verloren gehen können.
Jede elektronische Datendatei trägt im Namen das Datum, das Namenskürzel und das Experiment (The raw data files are named according to the following scheme):
Jahr-Monat-Tag_Namenskürzel_Experiment / year-month-day_initials_experiment
z.B. /e.g.: 2025-01-01_CRH_Adhesion assay
- the digital version/raw files should be on the file server with the date that can also be found in the lab book
- a printed version of the evaluated data should be attached in the lab book
For later use in publications it is important that we all stick to the same folder structure and naming of files. An example of such a user folder is given on the Fileserver and depicted on the next page:
Fileserver_LSH_Data/00_User/xUE_Universal_Example/
Data
Constructs
SuperSH2 agarose gels, sequencing results, plasmid map, etc.
Western Blots
Immunofluorescence staining
Flow Cytometry
ELISA/SEAP-Assays
.
.
Literature
Presentations
Thesis: for your thesis: for every figure in your thesis create a subfolder named with the Figure-Number and include in each subfolder both: the final figure as pdf- and illustrator-file and the used raw data (subdivided in panels A, B, etc.). The name of the raw data file should remain the same as it is found in your DATA-folder and lab book)
Laborarbeitsplatz
In den Laboren gibt es allgemeine Laborarbeitsplätze (z.B. in den beiden Zellkulturlaboren, im Infektionsraum oder der Bereich mit Proteingel-Elektrophorese) sowie private Arbeitsplätze, die zugewiesen sind. Arbeiten auf den privaten Arbeitsplätzen anderer soll nur nach Rücksprache erfolgen. Jeder ist für die Sauberhaltung seines Arbeitsplatzes, der dazugehörigen Regale und Schubladen selbst verantwortlich. Die Arbeitsfläche wird mindestens einmal pro Woche abgewischt und desinfiziert, die Schubladen/Regale sind regelmäßig abzustauben und ordentlich zu halten.
Zusätzlich hat jeder einen Lagerplatz in einem Kühlschrank bei 4°C, in einem Gefrierschrank/-fach bei -20°C und kann Proben in beschrifteten Boxen im -80°C-Schrank in Raum ML lagern. Für die Lagerung stehen Kunststoffboxen mit Deckel zur Verfügung, offenen Ständer (speziell für Eppendorfgefäße) sollen nur für Kurzzeit-Aufbewahrung (1 Tag) verwendet werden. Alle Proben (Eppis, Agarplatten, etc.) müssen mit Kürzel, Inhaltsbezeichnung und Datum beschriftet sein. Alle Lagerplätze sollen regelmäßig (mindestens 1x je Monat) aufgeräumt/aktualisiert werden. Bei Lagerung im -80°C Schrank ist für jede Box ein Datenblatt in den -80°C-Ordner abzulegen. Eine Vorlage für das Datenblatt findet sich auf dem Fileserver:
Fileserver_LSH/Vorlagen/Labor-Protokollierung/ -80C Box-Vorlage.doc
Das Öffnen von Tiefgefrierschränken bitte auf ein Minimum reduzieren (da sonst Vereisung und Fehlfunktion). Deshalb bereits VOR dem Öffnen überlegen, was entnommen werden soll und wo es zu finden ist (s. Datenblatt!!!). Allgemeine Laborarbeitsplätze sind nach jeder Benutzung zu säubern (z.B. sterile Werkbank, Proteingel-Elektrophorese, Abzug, Waagen, Geldoku) und alle verwendeten Lösungen, Apparaturen, etc. müssen wieder aufgeräumt werden.
Lab-Jobs
Jeder Mitarbeiter erhält einen Labjob, den er für die Allgemeinheit wahrnimmt. Der Lab-Job kann die Pflege einer allgemeinen Sammlung, die Verantwortung für ein Gerät oder die Aufsicht und Organisation eines Labors beinhalten. Darüber hinaus wird die Herstellung von Lösungen/Puffern etc. für die Allgemeinheit über Lab-Jobs. Die jeweils Verantwortlichen sind auf einer Liste am am Noticeboard am Eingang aufgeführt. Die Raum-Verantwortlichen organisieren auch die regelmäßige Pflege/Putzpläne für allgemeine Geräte wie sterile Werkbänke, Inkubatoren, etc.
Schließdienst
Der abendliche Schließdienst, der dafür sorgt, dass Geräte wie sterile Werkbänke, Vakkumpumpen, Geldoku oder die Propangasleitung ausgeschaltet sind, wechselt im wöchentlichen Turnus unter den wissenschaftlichen Mitarbeitern und Masterstudenten. Eine Liste für den abendlichen Kontrollgang des Schließdienstes hängt am Noticeboard am Eingang.
Computernutzung
Im Seminarraum befindet sich ein Computer zur allgemeinen Nutzung. Bitte jederzeit die Nutzung für dienstliche Zwecke ermöglichen. Private PCs/Notebooks können an das Netzwerk angeschlossen werden. Voraussetzung ist ein aktueller Virenscanner und die Aktivierung einer Firewall. Den Zugang zu den Netzwerk-Speichern des Lehrstuhls im Rechenzentrum (Fileserver_LSH und Fileserver_LSH_Data) richtet Christof ein. Auf dem Fileserver_LSH_Data/02_SOFTWARE stehen verschiedene Software-Programme zur Verfügung, darunter Adobe Photoshop CS4, Adobe Acrobat Pro9, Adobe Illustrator CS4, MS Office 2016, Clone Manager 9, Endnote X9, SigmaStat 4.0, Leica LAS, Biorad QuantityOne, VarioScan SkanIt. Um den problemlosen Austausch der Daten innerhalb des Labors zu gewährleisten, müssen die oben angegebenen Versionen dieser Programme für alle dienstlichen Zwecke verwendet werden bzw. die Daten in einer mit diesen Programmversionen kompatiblen Form gespeichert werden.
Die Mitarbeit jedes Einzelnen ist gefragt
Jeder Mitarbeiter kann durch wenig Aufwand dazu beitragen, dass sich die Zusatzarbeiten im Labor für Alle minimieren. So soll jeder durch vorausschauende Planung den anfallenden Verbrauch/Abfall/Aufräumaufwand möglichst gering halten und auch bei der Entsorgung von Abfällen mithelfen. Bitte Autoklaviersäcke nicht bis zum „Überquellen“ füllen, sondern rechtzeitig verschließen, in der Wanne deponieren und neuen Autoklaviersack in den entsprechenden Müllbehälter geben.
Jeder ist auch selbst für das Befüllen der Boxen mit Pipettenspitzen und das Auffüllen von Eppis verantwortlich. Autoklaviergut wird in der Plastikkiste gesammelt und von Claudia/Susanne für alle autoklaviert.
Zum verträglichen Umgang gehört auch das Verhalten und die sauberkeit in der Teeküche und im Sozialraum. Wenn ich im Verlaufe von 1 Monat nie die Spülmaschine ein- oder ausgeräumt oder die Kaffeemaschine aufgefüllt habe, dann habe ich mich sehr wahrscheinlich auf Kosten von Anderen am sauberen Geschirr bedient.
Chemikalien/Lösungen
Viele Lösungen werden als Lab-Job angesetzt und entweder als Stocklösung an festgelegten Plätzen für Alle zugänglich aufbewahrt oder als aliquotierte Lösung bereitgestellt, wobei man sich bei Bedarf ein Aliquot an den eigenen Arbeitsplatz mitnimmt und dort für die weitere Verwendung aufbewahrt. Weitere Lösungen für den eigenen Bedarf sind selbst anzusetzen (bitte die benötigten Mengen im Voraus abschätzen und nicht Liter-weise Lösungen ansetzen, wenn man für zwei Experimente insgesamt nur 10 ml braucht) und korrekt beschriftet (Kürzel, Datum, Inhalt: Bezeichnung und Konzentration; Gefahrensymbol) am eigenen Arbeitsplatz aufzubewahren. Lösungen und Aliquots am Arbeitsplatz der Kollegen NUR nach Rücksprache benutzen. Stocklösungen, aliquotierte Lösungen und die jeweiligen Verantwortlichen sind auf der Liste am Schwarzen Brett „Lab-Jobs“ aufgeführt. Für das Ansetzen von Lösungen gibt es ein Rezeptbuch, das als Datei auf dem Fileserver bereit liegt:
Fileserver_LSH//Rezepte/ Rezepte-2025.doc
Allgemeine Sammlungen
Der Lehrstuhl unterhält verschiedene Sammlungen, die eine wichtige Ressource für Alle darstellen und mit denen deshalb besonders umsichtig und verantwortlich umgegangen werden muss.
Stammsammlung: umfasst alle Bakterienstämme, die nach Nummern abgelegt sind. Lagerung bei -70°C in Truhe auf Ebene 5 sowie in Raum ML6 :
– nichtpathogene Bakterien (=gleichzeitig Repositorium aller hergestellten Plasmide)
– pathogene Bakterien und Mutanten (Schlüssel dafür bei Christof)
Die Stammsammlung ist auch als MS Access-Datei vorhanden. Jeder neu hergestellte oder von extern erhaltene Bakterienstamm erhält eine neue Stammnummer und wird in der Stammsammlung eingefroren sowie elektronisch erfasst. Die Verteilung der Nummern erfolgt über Christof. Zugang zur Stammsammlung haben nur Susanne und Christof.
Plasmidsammlung: umfasst alle isolierten Plasmide aus den nichtpathogenen Bakterienstämmen der Stammsammlung. Die Numerierung ist identisch mit der Stammsammlung (Lagerung im allg. Kühlschrank bei 4°C)
Zellsammlung: umfasst Stocks aller Zellinien und primärer Zellen. Werden im flüssigen Stickstoff in der Medienküche aufbewahrt. Die Zellsammlung wird von Susanne verwaltet.
Virensammlung: umfasst alle lentiviralen, adenoviralen, AAV-Partikel und Bakteriophagen. Werden bei -70°C aufbewahrt. Access-Datei!
Oligosammlung: umfasst alle Oligos, die für Klonierungen, PCRs, etc. bestellt wurden. Die Oligos werden als 10 mM Stammlösung in TE-Puffer bei -20°C in der allgemeinen Sammlung, nicht in privaten Gefrierschränken, aufbewahrt. Bestellung neuer Oligos und Ablage der Informationen erfolgt über Jan Kuiper.
Antikörper-Sammlung: Sammlung aller Primärantikörper (kommerziell oder selbst hergestellt) und Sekundärantikörper (Enzym- oder Fluoreszenz-konjugiert). Datenblätter werden im Ordner „Antikörper“ gesammelt. Claudia Hentschel organisiert die Antikörper-Sammlung.
Protein-Sammlung: Hier werden alle rekombinanten, gereinigten Proteine erfasst.
Inhibitor-Sammlung: Hier werden Informationen zu allen pharmakologischen Inhibitoren und die Konzentration, das Lösungsmittel und der Aufbewahrungsort von Stammlösungen abgelegt. _______________ organisiert die Inhibitor-Sammlung.
Enzym-Sammlung: umfasst alle kommerziellen und selbst hergestellten (DpnI) Restriktionsenzyme, die bei -20°C gelagert werden. Entnahme von Enzymen aus den Vorratsgefäßen nur mit FILTERSPITZEN!
Die Enzyme dürfen nur für eine möglichst kurze Zeitspanne (1-2 Minuten) aus dem Gefrierschrank entnommen werden (d.h. direkt vor Gebrauch) und werden während dieser kurzen Zeit in einem auf -20°C-gekühlten Paraffinblock gelagert.
Ordner mit Dokumentation zu allen Sammlungen finden sich in den Regalen am allgemeinen Arbeitsplatz im Bürobereich.
Kompetente Zellen
Aliquots kompetenter Bakterien werden bei -80°C gelagert. Je Aliquot sind 200 µl weggefroren, ausreichend für 2 Transformationen. Verschiedene Stämme stehen für unterschiedliche Zwecke zur Verfügung. Der Standardklonierungsstamm ist NovaBlue. Bei der Entnahme möglichst zügig vorgehen, Erwärmung der Aliquots führt zu verringerter Kompetenz, nicht mehr Aliquots entnehmen als tatsächlich gebraucht werden.
Enzyme
Polymerasen, Restriktionsenzyme, etc. werden bei -20°C in Boxen bzw. auf Paraffinblöcken gemeinsam mit den entsprechenden Puffern aufbewahrt. Diese Enzyme werden von Allen gemeinsam benutzt. Deshalb besonders reinlich mit den Enyzm-Stammlösungen umgehen und Verunreinigungen (Filterspitzen !!!) sowie Erwärmung vermeiden.
Bei der Entnahme von Enzymen, die Eppis mit der Stammlösung ausschließlich in einem auf -20°C-gekühlten Paraffinblöckchen zum Arbeitsplatz transportieren und darin zum Pipettieren belassen. Auch mit Paraffinblock möglichst das Enzym nur kurzzeitig aus dem -20°C Gefrierschrank entnehmen (< 2 Minuten) und sofort nach dem Pipettieren wieder zurückbringen. Beim Öffnen der Vorratsgefäße auf jeden Fall Kontakt der Deckelinnenseite mit der Haut vermeiden!!
Bestellungen
Wenn allgemeine Chemikalien oder Einwegartikel zur Neige gehen, RECHTZEITIG (d.h. vor dem Aufbrauchen der letzten Packung oder dem Abpipettieren des letzten Mikroliters) Bescheid geben: entweder Anschreiben an die Tafel im Labor oder den Bestellern sagen. Projektspezifische Chemikalien/Antikörper etc. oder Laborutensilien/-geräte vor Bestellung mit Christof absprechen.
Für Oligobestellungen über Jan die Oligo-Informationen in die Bestell-Liste auf dem Fileserver eintragen: Fileserver_LSH:\Stammsammlung\Primer\01_PrimerOrdering_Primerbestellung
Andere on-line Bestellungen mit Christof absprechen.
Bestellung von Sequenzierungen über pre-paid labels von LGC oder GeneWiz. Bitte nicht mehr als 2 Sequenzierung pro neuem Konstrukt. Größeren Sequenzierbedarf mit Christof absprechen. Lieferungen von Reagenzien sollen dem Besteller übergeben werden. Besteller prüft auf Vollständigkeit (unter Nutzen des Lieferscheins) und bestätigt mit Datum und Unterschrift. Der Lieferschein und eventuell beiliegende Rechnungen werden im Sekretariat an Petra Schnurr weitergegeben. Bei Lieferung von Chemikalien, Antikörpern, Enzymen und anderen Reagenzien werden die Datenblätter mit einem Datum versehen und in den entsprechenden Ordnern abgelegt (s. „Allgemeine Sammlungen“). Auf den Datenblättern wird eventuell das Lösungsmittel, die Konzentration und eventuell abgefüllte Aliquots sowie der Lagerort vermerkt.
ALLE Chemikalien, Reagenzien, Enzyme, etc. werden an zugänglichen und bekannten Orten gelagert.
Laborsicherheit
Siehe dazu Safety Guidelines